Der vollautomatisierte Extraktionsautomat MagCore Plus II besticht durch seine Aufreinigung in höchster Qualität und der Vielfalt an Kits.
DNA/RNA kann aus einer Vielzahl an Ausgangsmaterialien extrahiert werden: Blut, Plasma, Körperflüssigkeit, Zellen, Gewebe, FFPE, Bakterien, Viren, forensische Proben.
Neben viraler, bakterieller und genomischer DNA erhält man auch spezielle Endprodukte wie zellfreie DNA (ct DNA) oder miRNA (micro RNA).
Und seit 2022 können wir alle Extraktionsautomaten und Kits unter der IVDR Zertifizierung anbieten !
Nachfolgend sehen Sie eine Übersicht der verfügbaren Kits mit Angaben der Kitgrößen und dem validierten Probenmaterial.
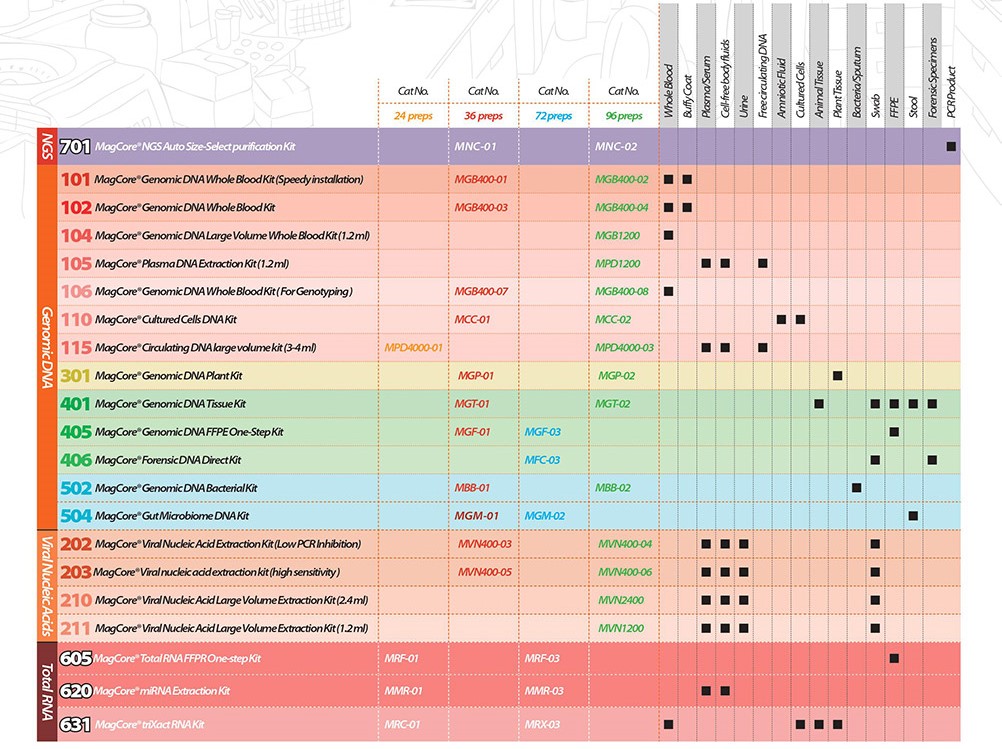
….. und es werden laufend neue Kits entwickelt und das Portfolio erweitert.
Der einzigartige erste vollautomatische Grössenaufreinigungs -Kit für NGS Proben wurde bereits in einem früheren Blog vorgestellt.
Zusätzliche Informationen finden Sie in folgendem Flyer und Video:
Mit dem BioProducts Wandplaner 2024 den Überblick bewahren.
Wir haben auch für das kommende Jahr wieder einen Wandplaner gestaltet und sind auch ein bisschen stolz darauf. Es würde uns sehr freuen, wenn er wieder einen Platz in ihrem Labor oder ihr Büro findet.
Gerne legen wir Ihnen auf Wunsch bei Ihrer nächsten Bestellung ein druckfrisches Exemplar des Wandplaners oder Tischkalender bei oder senden Ihnen die neuen Exemplare zu.
Es ist wieder soweit, der BioProducts Tischkalender 2024 ist bei uns eingelangt. Es würde uns freuen, wenn er sie im kommenden Jahr wieder bei ihrer Terminplanung unterstützt.
Wie gewohnt legen wir den druckfrischen BioProducts Tischkalender 2024 bei ihrer nächsten Bestellung bei oder senden Ihnen auf Wunsch ein Exemplar zu.
Zusätzlich können wir unseren beliebten BioProducts Wandplaner 2024 bereits ankündigen, der in Kürze verfügbar sein wird.
Dermatophyten befallen hauptsächlich Haut, Nägel und Haare. Da Dermatophyten-Infektionen in der Regel keine schwerwiegenden gesundheitlichen Komplikationen verursachen, suchen Betroffene oft keine medizinische Behandlung auf. Jedoch können auch Nicht-Dermatophyten Pilze Haut, Nägel und Haare befallen und ähnliche Symptome aufweisen.
Warum ist es wichtig Dermatophyten und andere Pilze bei Infektionen zu bestimmen und Gegenmaßnahmen einzuleiten ?
Für eine rasche Diagnose der Infektion und ein Erkennen einer multiplen Infektion sollte man ein Multiplex PCR-Panel einsetzen.
| Artikelnummer | Bezeichnung |
| HP84115 | Dermatophytes and other Fungi, 12-well |
Es gibt 3 Pilz Gattungen, die Infektionen verursachen und wegen Ihrer Fähigkeit Keratin zu verwerten als Dermatophyten klassifiziert werden.
Nannizzia gypsea wird auch als Dermatophyte gesehen. Vor kurzem noch zählte diese Spezie zu der Dermatophyten Gattung Microsporum (Microsporum gypseum), jedoch haben Mykologen, die sich mit Systematik beschäftigen eine neue Gattung hervorgerufen. N. gypsea kommt natürlich in Böden vor, kann aber zu Infektionen beim Menschen führen.
Neben den klassischen Dermatophyten können auch Schimmelpilze und Hefen Fingernägel und vor allem Zehennägel befallen. Schimmelpilze der Gattung Aspergillus sind omnipräsent und ihre Sporen findet man in Innen und Außenräumen. Es gibt eine Vielzahl von Aspergillus Spezies, die Infektionen hervorrufen können, A. flavus , A terreus bzw A. niger werden jedoch am häufigsten diagnostiziert.

Scopulariopsis Spezies kommen oft zusammen mit Dermatophyten Infektion vor, können aber auch der Erst- und Hauptverursacher von Nagelinfektionen sein. Scopulariopsis ist schwer zu behandeln, deswegen ist eine frühe Diagnose wichtig. Scopulariopsis brevicaulis ist die am häufigsten gefundene Spezies einer Gattung, die natürlich in Böden als Saprophyt vorkommt.
Auslöser für Hautinfektionen können auch Spezies der Gattung Candida sein. Candida Pilze sind Hefe, die bei den meisten Menschen als unschädlicher Mitbewohner der Schleimhäute leben.
Candida Infektionen können z.B. entstehen, wenn die Bakterienflora auf der Haut durch die Einnahme von Antibiotika zerstört ist.
Candida sind auch ein Teil des Mikrobiom des Darms. In Ausnahmesituationen (wie z.B Immunschwäche) kann sich der Pilz jedoch ungehindert vermehren und er schwächt den Körper, kann sogar bei Befall der Blutgefässe zur tödlichen Gefahr werden (Sepsis).
Candida albicans ist die Spezies, die am häufigsten bei Infektionen gefunden wird. C. guillermondii , C. glabrata und C. parapsilosis können auch Infektionen hervorrufen.
Das Dermatophyten Panel läuft auf dem Multiplex Tandem PCR System von Ausdiagnostics und es sind noch viele andere Diagnostik-Panel verfügbar. Eine Übersicht der Panel finden Sie in folgendem Flyer:
Unser HybriSpot-System bietet eine vollautomatisierte Lösung für Syndromic Panel Testing. Es basiert auf einer innovativen Kombination von Multiplex-PCR, Macroarray Chips und der innovativen DNA Flow Technologie, was schnelle, sehr sensitive und genaue Ergebnisse ermöglicht.
Das System zeichnet sich durch eine einfache Handhabung aus:
Ein weiterer Pluspunkt ist, dass sehr viele Ausgangsmaterialien mit dem HybriSpot-System validiert sind: abhängig von jeweiligen Test können verschiedenste Abstriche, Liquid-based cytology (LBC)-Proben, Paraffin-eingebettetes Gewebe (FFPE), Blutkulturen, bronchoalveoläre Lavage (BAL) und Vieles mehr untersucht werden.

Für das HybriSpot-System ist eine umfangreiche Palette von Paneltests, die auf der DNA Flow-Technologie basieren, verfügbar:

Das HybriSpot-System erlaubt maßgeschneiderte Lösungen je nach Größe und Probenaufkommen des Labors: vom Gerät für die manuelle Durchführung bis zu vollautomatischen Lösungen.

Wenn Sie den HybriSpot 12 PCR Auto „in Aktion“ sehen wollen, dann laden wir Sie sehr herzlich zu unserem BioProducts-Workshop am 07.09.2023 bei BioProducts in Stockerau ein! Bei Interesse bitten wir um eine Anmeldung per E-Mail.
Um hohe Qualitätsstandards zu gewährleisten unterzieht sich BioProducts jährlich einem Audit eines zertifizierten externen Auditors. Unser Bemühen zur kontinuierlichen Qualitätsverbesserung wurde durch den Erhalt des Zertifikats ISO 13485:2016 bestätigt.
Unsere Qualitätspolitik und Werte
Von 8.-10. Mai 2023 fand das 26. Dosch-Symposium, mit dem Thema: Hygiene und Infektionen in Einrichtungen des Gesundheitswesens, in Velden am Wörthersee statt.
Wir waren mit einem eigenen Stand vertreten, bei dem wir unsere innovativen Produkte für die medizinische Diagnostik vorstellen durften.



Wir bedanken uns bei den Veranstaltern für die gelungene Zusammenarbeit.
Es gibt wohl wenige PCR-Tests auf der Welt deren Nutzen so offensichtlich sind wie bei der Hereditären Hämochromatose. Denn die Behandlung kostet nichts - ganz im Gegenteil! Regelmäßiges Blutspenden reicht um die gefürchtete Leberzirrhose gänzlich zu verhindern. Eine „Win-Win-Situation“ für alle. Obwohl wenige Genloci reichen um die Diagnose zu sichern, wird die Testung meist nur zur Bestätigung einer Schädigung der Leber durch die Eisenspeicherkrankheit verwendet und das obwohl wir heute wissen, dass die HH die häufigste angeborene Stoffwechselerkrankung in der kaukasischen Bevölkerung ist. Laut AWMF Leitlinien sollte jeder Patient mit auffälligen Laborparametern (erhöhte Transferrin und Ferritinsättigungen) genetisch abgeklärt werden, sowie alle Familienmitglieder 1. Grades.
Unser Angebot für den Nachweis der klinisch relevanten Mutationen im HFE-Gen basiert auf verschieden PCR-Plattformen und lässt sich daher leicht im Labor mit bereits vorhandener Ausstattung etablieren.
Wir unterstützen Sie bei der Austestung und Etablierung mit unserem molekularbiologischen Service vor Ort und bieten Ihnen attraktive Preise für eine Evaluierung. Auch können wir entsprechende Geräte zu Testung bereitstellen.
Vertrauen Sie auf unsere Arbeit in der PCR-Diagnostik seit über 20 Jahren.
Wir freuen uns Ihnen unsere neue Lösung zur automatisierten Abarbeitung von Hybridisierungstreifen (Line Probe Assays) vorstellen zu dürfen:
Das Gerät Dynablot Heat VL ist für die Hybridisierung von Streifen mit PCR-Amplifikaten geeignet. Der Hybridisierungsautomat besteht aus drei separaten Heizblöcken, welche jeweils ein Fassungsvolumen von 10 Streifen haben. Das heißt es können bis zu 30 Streifen gleichzeitig bearbeitet werden. Das Gerät ist mit allen gängigen Streifentests kompatibel.

Der Hybridisierungsautomat arbeitet in einem breiten Temperaturbereich von 35-65 °C und verfügt außerdem über zwei Plätze zum Beheizen von Reagenzien (mit eingebautem Magnetrührer). Über die bis zu 6 Pumpen werden die verschiedenen Reagenzien zu den jeweiligen Streifen befördert. Das Gerät bietet einige praktische Merkmale, so kann z.B. die gesamte Front zum Beladen der Reagenzien herausgezogen werden (siehe Bild oben).
Es handelt sich hierbei um ein kompaktes Laborgerät mit den Abmessungen 60x44x48 cm. Um eine einfache Bedingung zu ermöglichen, ist das Gerät zudem mit einem modernen Touchscreen versehen. Der Dynablot Heat VL wird mit einer großen Waste-Bottle geliefert. Ein Overflow der Waste-Bottle wird durch einen eigenen Sensor verhindern.

| Artikelnummer | Bezeichnung |
| 905-01 | Dynablot Heat VL |
Vertrauen Sie auf unsere über 20-Jährige Erfahrung beim Geräteservice und -support im Diagnostikbereich.
Der HPV Direct Flow Chip Test kann 35 HPV Genotypen bestimmen. Der Test basiert auf PCR mit anschliessender DNA Hybridisierung und automtischer Auswertung der Chips mittels inkludierter Software. Alle Schritte laufen voll automatisiert im HS12A Gerät ab .

Neben DNA Extrakten ist das System auch hervorragend etabliert für Proben ohne Extraktion, z.B. von Cervixabstrichen (PAP-Abstriche), LBC Proben (Liquid-Based Cytology) oder von FFPE Geweben. Nach nur wenigen Vorbereitungsschritten wird das Ausgangsmaterial zum lyophilisierten PCR Mix dazugegeben und alle weiteren Schritte bis zur Diagnose erfolgen im HS12A Gerät.

Neben dem HPV Panel können noch andere Tests auf dem vollautomatisierten HS12A System eingesetzt werden. Aktuell sind folgende Tests verfügbar:
| Tick-Borne Bacteria Flow Chip | Gleichzeitiger Nachweis von krankheitserregenden Bakterien, die von Zecken übertragen werden: Rickettsia, Rickettsia Typhus Group, Rickettsia Spotted Fever Group, Erhlichia, Anaplasma, Francisella, Bartonella, Borrelia, Coxiella |
| STD Direct Flow Chip | Gleichzeitiger Nachweis von 12 STD (sexuell übertragbare Krankheiten) verursachenden Pathogenen |
| Sepsis Flow Chip | Gleichzeitige Detektion von 36 Bakterienarten, Pilzen und 20 Antibiotikaresistenzgenen |
| AMR Direct Flow Chip | Gleichzeitige Detektion von 20 Antibiotikaresistenzgenen |
| Bacterial CNS Flow Chip | Gleichzeitiger Nachweis von Bakterien und Pilzen, die Infektionen im Zentralennervensystem (CNS) verursachen. Mit DNA aus Cerebrospinalflüssigkeit (CSF) |
| Respiratory Flow Chip | Gleichzeitiger Nachweis der Hauptverursacher von respiratorischen Krankheiten (u.a. SARS-CoV-2, Influenza, RSV, Parainfluenza, Bordetella) |
In der molekularen Diagnostik werden momentan hauptsächlich Short Read Sequenzierer von den Firmen Illumina bzw. Thermo Fisher eingesetzt weil ihre Technologie sehr verlässlich ist und weniger Fehler produziert als die nächste Generation der Long Read Sequenzierer (PacBio, Oxford Nanopore). Der Nachteil der Short Read Sequenzierungen ist, dass die Vorbereitung relativ aufwendig ist.
Bei der Vorbereitung der Probe, auch "library preparation" genannt, sind mehrere Schritte für eine erfolgreiche Sequenzierung nötig und um qualitativ gute Ergebnisse zu erhalten sollte die library preparation sorgsam und unter höchsten Standards gemacht werden. Folgende Schritte werden benötigt:
Warum brauchen wir eine Größenauswahl ?
–> ein Problem, das Sequenzierer der dritten Generation zu lösen versuchen.
Welche Methoden zur Grössenauswahl gibt es ?
Enzymatisch
z.B Nextera Kits von Illumina. Benutzen Transposons zur Fragmentierung. Ergebnisse aber oft unzufriedenstelllend (Breite Grössenverteilung)
Gel Elektrophorese
Sehr genaue Methode aber extrem aufwendig und zeitintensiv
Magnetic bead – Technologie
DNA-Fragmente der gewünschten Größe binden in Gegenwart des optimierten Puffers an die Beads. Ein Magnet wird verwendet, um die Beads/DNA zu befestigen Kostengünstig, weniger zeitaufwendig als Gel Elektrophorese, gute Qualität
Eigenschaften:
1. Automatische DNA Aufreinigung und Grössenselektion in ~ 30min
2. "magnetic bead" Technologie
3. Hohe Ausbeute

4. Wenig Arbeitszeit
5. Konstant gute Qualität – keine manuellen Fehler
Anwendungen:
1. DNA library peparation für NGS
2. Aufreingungen für PCR, Hybridisierungen oder Real-Time PCR
Datum: 30. November. 2022
Ort: FLEMINGS CONFERENCE HOTEL, WIEN
Neubaugürtel 26-28,
A-1070 Wien
Das Programm sowie eine Bildergalerie finden Sie hier.
Digitale PCR ist eine spezielle Weiterentwicklung der klassischen PCR. Das Besondere an dieser PCR Anwendung ist, dass einzelne DNA Moleküle in winzigste Kompartimente getrennt werden, sodass jeweils nur 1 Strang oder keiner in einem Kompartiment amplifiziert wird. Die Art der Aufteilung hängt von der Technologie ab – es wird grob zwischen digital droplet PCR und chamber digital PCR unterschieden. In einem Fall (ddPCR) werden kleinste Öl-Tropfen getrennt und in dem anderem Fall Moleküle in kleinste Mikrobereiche aufgeteilt (cdPCR).
Digitale PCR besticht durch hohe Sensitivität und Spezifität und durch die hohe Verdünnung ist die Beeinträchtigiung durch PCR Inhibitoren fast ausgeschlossen.
Wir sind immer an innovativen Produkten interessiert und können jetzt auch erstmals ein Produkt basierend auf dPCR anbieten.
Die Quantifizierung von BCR-ABL1 bzw. ABL1 aus Vollblut von Patienten mit positiver chronischer myeloischer Leukämie (CML) ist sehr gut geeignet für digitale PCR. Der Dr. PCR™ BCR-ABL1 Major IS Detection Kit ist ein in-vitro-Diagnose Kit und soll das Verhältnis von BCR-ABL1 zu ABL1 messen. Der Kit erlaubt eine akkurate Beobachtung von Leukämie Patienten während einer Therapie bezüglich Verhältnis BCR ABL1 - ABL1 Transkripte.

Der Kit besteht aus 1 Tube dRT-PCR Master Mix und 1 Tube Primer & Probe Mix. Die beiden Reagentien werden in einem Reaktionsgefäß mit dem RNA Extrakt zusammengemischt und in eine mitgelieferte Kartusche injiziert. In der Kartusche wird die Probe in 22000 Kompartimente (50nm) verteilt. Diese Kartusche wird in das real-time Gerät plaziert und nach der qPCR wird das Ergebnis ausgegeben.
Fakten
Ringversuche dienen dazu, die Leistungsfähigkeit eines Labors und ihres Testsystems zu überprüfen. Wenn man medizinische Proben analysiert, muss man sichergehen, dass die Testergebnisse stimmen. Bei Ringversuchen werden identische Proben an teilnehmende Labore verschickt. Es gibt verschiedenste Einflussfaktoren, die die Testergebnisse beeinflussen, wie zum Beispiel die Laborausstattung, die Arbeitsweise der Mitarbeiter und natürlich das Testsystem. Die Testergebnisse werden vom Ringversuch-Anbieter gesammelt und statistisch verarbeitet, um die Messgenauigkeit der Labore zu bewerten
Wir von BioProducts überprüfen unsere Testsysteme mittels Ringversuchen um den Kunden zuverlässige Analysen zu garantieren.
Im Jahr 2022 konnten wir bisher nachweisen, dass unsere HPV Testsysteme den Qualitätsstandards entsprechen. Beim HPV Zervixkarzinom Ringversuch haben wir mit unserem Extraktionssystem von RBC (MagCore® Genomic DNA FFPE One-Step Kit) und dem Panelsystem von Ausdiagnostics (High-Risk HPV Genotyping 8-well (Ref 23201) erfolgreich teilgenommen. Der molekularbiologische Nachweis zur Identifizierung und Unterscheidung der HPV Typen wird immer wichtiger zur Früherkennung von Krebserkrankungen. Es gibt mittlerweile mehr als 120 bekannte HPV Typen und PCR Tests können zuverlässig unterscheiden ob es sich um einen „High Risk“ oder „Low Risk“ Typ handelt. Infektionen mit Hochrisiko-HPV-Typen können zu Tumoren (z. B. Gebärmutterhalskrebs bei HPV16/HPV18) führen, während Niedrigrisiko-HPV-Typen ( z.B. HPV6 und HPV11) für Genitalwarzen verantwortlich sein können.
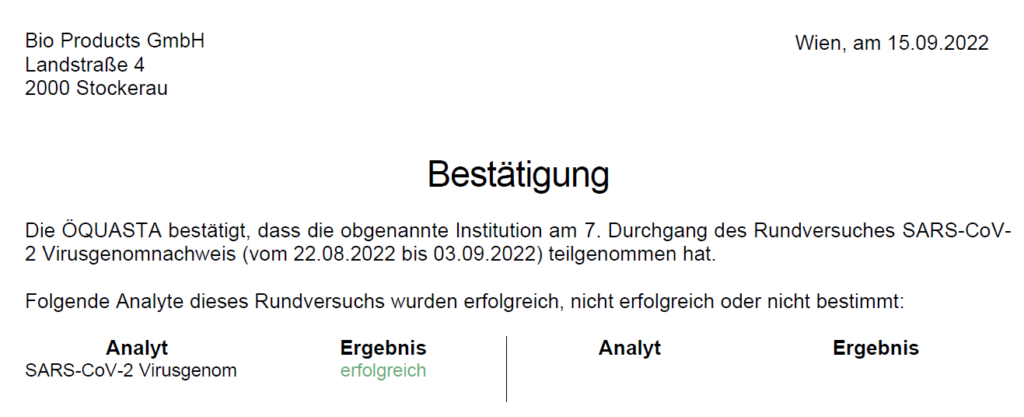
Weiters überprüfen wir regelmäßig die Qualität unserer SARS Cov-2 Testsysteme bei den ÖQUASTA (Österreichische Gesellschaft für Qualitätssicherung und Standardisierung medizinisch-diagnostischer Untersuchungen) Rundversuchen. Sowohl unser POC Analysegerät (LR 1000) als auch das respiratorische Panel (SARS-CoV-2, Influenza & RSV 8-well) von Ausdiagnostics haben 100% der Proben beim letzten Rundversuch (7; August 2022) richtig analysiert. Das gelang nur 73% aller Teilnehmer.
Am 29.09. und 30.09. September 2022 findet die 21. ÖGH (Österreichiche Gesellschaft für Humangenetik) Jahrestagung im Zentrum von Graz statt.
Die ÖGH Jahrestagung ist die wichtigste wissenschaftliche Veranstaltung im Bereich der Humangenetik in Österreich. Die österreichischen Humangenetik-Institute präsentieren die neuesten Trends in der Forschung und Entwicklung mit Schwerpunkt Genom-Diagnostik. Führende Experten werden bei dem Treffen diagnostische Möglichkeiten und Probleme, die die neuesten Technologien in der Genomforschung bringen, diskutieren
Lernen sie die neuesten Errungenschaften in der Humangenetik-Forschung und Diagnostik kennen und besuchen sie in den Pausen unseren Stand. Wir stehen zur Verfügung um mit Ihnen über aktuelle Trends in der Diagnostik zu sprechen und wir bemühen uns Lösungen für offene Fragestellugen anzubieten.
Themen:
Genomsequenzierung, Single-Cell Analysen, Mutationsanalysen, Epigenetik, Tumor und cfDNA, ctDNA und vieles mehr
Wann und wo ?