
Die Ursache bei mehr als 90% der Patienten mit einer chronischen myeloischen Leukämie (CML) und etwa 20% der Patienten mit akuter lymphatischer Leukämie (ALL) ist das Vorhandensein eines sogenannten Philadelphia-Chromosoms. Beim Philadelphia Chromosom handelt es sich um ein verkürztes Chromosom 22. Bereits im Jahr 1960 entdeckten die Wissenschaftler Peter Novell und David Hungerford mittels Mikroskopie von Zellen, dass 95% aller CML Patienten dieses verkürzte Chromosom 22 haben. Die Konsequenz dieser Chromosomen Anomalie ist eine starke Vermehrung von Leukozyten (weißen Blutkörperchen), speziell von Granulozyten und ihren Vorstufen, sowohl im Blut als auch im Knochenmark. Im darauffolgenden Jahrzehnt wurde dann festgestellt, dass bei der Entstehung des Philadelphia-Chromosoms es zu einem Austausch von Chromosomenstücken zwischen Chromosom 22 und 9 kommt. Das Philadelphia-Chromosom kann durch Diagnosemethoden wie z.B. G-Banding oder FISH nachgewiesen werden.
Dieses Chromosom ist auch jenes, das CML bedingt. Es kommt zu einer Fusion vom Gen BCR auf Chromosom 22 mit dem Gen ABL1, wobei das gebildete Fusionsprotein BCR-ABL1 durch unkontrollierte Tyrosinkinase-Aktivität eine Vermehrung der Leukämiezellen bewirkt.
Das BCR-ABL-Fusionsgen bildet sich in verschiedenen Varianten. Die unterschiedlich großen Fusionsproteine werden nach ihrer Molekülmasse beschrieben. Die häufigste Form ist die p210 (>90%), während p190 (~5%) und p230 (<1%) bzw. andere Formen selten vorkommen.
Während der Therapie ist es wichtig den Anteil des jeweiligen Fusionsproteins zum unveränderten ABL1-Protein nachzuverfolgen. Dazu werden die absoluten Mengen der BCR-ABL1 und ABL1-Transkripte durch eine quantitative Realtime-PCR bestimmt. Wir bieten dazu einen Kit zur Quantifizierung der Variante p190 (e1a2) im Verhältnis zum unveränderten ABL1-Transkript (Art.nr. RQ-115-6M) und einen Kit zur Quantifizierung der Variante p210 (b3a2 und b2a2) im Verhältnis zum ABL1-Transkript (Art.nr. RQ-105-6M) an.
| Protein | Variante | Bruchpunkt (BCR-Region) |
| p210 | Major (M) | Exon 13 (b2a2) |
| p210 | Major (M) | Exon 14 (b3a2) |
| p230 | micro (μ) | Exon 1 (e1a2) |
| p190 | minor (m) | Exon 19 (e19a2) |
| Artikelnummer | Bezeichnung |
| BP101 | BioPro BCR-ABL1 M, m, micro Multiplex, 24 Tests |
| BP011 | BioPro BCR-ABL Control RNA - b3a2, 100 µl |
| BP012 | BioPro BCR-ABL Control RNA - b2a2, 100 µl |
| BP013 | BioPro BCR-ABL Control RNA - e1a2, 100 µl |
| BP014 | BioPro BCR-ABL Control RNA - e19a2, 100 µl |
| RQ-115-6M | BCR-ABL p190 One-Step Realquality, 100 Tests |
| RQ-116-SM | BCR-ABL p190 Standards, 5 x 135 µl |
| RQ-105-6M | BCR-ABL p210 One-Step Realqualitiy, 100 Tests |
| RQ-54-SM | BCR-ABL p210 Standards, 5 x 135 µl |
Pneumonie beschreibt eine Infektion der Atemwege, die durch unterschiedliche Erreger ausgelöst werden kann und durch verschiedenste Symptome begleitet wird.
Eine Pneumonie bzw. Lungenentzündung kann milde bis schwerwiegende Symptome mit sich bringen.
Die Infektion kann bis zum Tod führen. Vor allem Kinder, ältere Menschen, Menschen mit Vorerkrankungen und immunsupprimierte Personen sind besonders gefährdet. Um dem Vorzubeugen kann eine PCR Analyse in wenigen Stunden den Erreger nachweisen – handelt es sich um eine bakterielle Infektion kann eine Antibiotikabehandlung eingeleitet werden.
Atypische und typische Pneumonien unterscheiden sich durch ihre Biologie und ihre Behandlung.
Atypische Bakterien haben besondere Eigenschaften (z. B. keine Zellwand, intrazellulär) und benötigen oft eine spezialisierte Behandlung wohingegen typische Bakterien gut kultivierbar sind und eine Zellwand besitzen, die sie leichter mit Standardantibiotika behandelbar machen.
Das Pneumonie Panel am High Plex System
Eine ideale Lösung für eine schnelle und umfangreiche Diagnostik von atypischen und typischen Pneumonien ist unser Pneumonia-Panel, das auf Multiplex PCR basiert, vollautomatisiert durchgeführt wird und das gleichzeitig 12 Bakterien und 4 Pilzarten nachweisen kann.

Zusätzlich bieten wir ein Atypisches Pneumonia Panel an, das nur die atypischen Erreger erkennt und dadurch schneller abgearbeitet werden kann.
| Artikelnummer | Bezeichnung |
| HP20632 | Atypical Pneumonia, 8-well |
| HP20631 | Pneumonia, 16-well |
Atypische Pneumonien
Verschiedene Erreger wie zum Beispiel Chlamydophila pneumoniae, Mycoplasma pneumoniae und Legionella Spezies zeigen mildere respiratorische Symptome, aber bringen oft extrapulmonale Nebeneffekte wie zum Beispiel Kopfschmerzen, Muskelschmerzen und gastrointestinale Probleme mit sich.
Atypische Bakterien sind schwieriger zu behandeln, weil ß-Lactam Antibiotika, die gut bei Typischen Pneumonien einsetzbar sind, nicht wirken. Der Grund ist, dass Atypische Bakterien keine Zellwand haben oder intrazellulär leben. Hier muss man Antibiotika einsetzen, die in Zellen eindringen können, wie zum Beispiel Makrolide, Tetracycline oder Fluoroquinolone.
Weil Infektionen mit Atypischen Pneumonien gewöhnlich nicht in Infektionswellen, sondern sporadisch bei immunsupprimierten Personen auftreten ist eine exakte Diagnostik essentiell.
Legionelle Infektionen betreffen gewöhnlich ältere Personen bzw. Neugeborene. Die Ansteckung erfolgt nicht über Personenkontakt sondern kann zum Beispiel über "verschmutztes" Wasser bzw. Gartenkompost passieren. Infektionen mit Legionellen (Legionärskrankheit (Legionellose)) können zum Tod führen !
Und eine umgehende Behandlung ist empfehlenswert, weil Infektionen mit atypischen Bakterien eventuell auch zu Veränderungen der Herzkranzgefäße führen können bzw Multiple Sklerose begünstigen. Weiters vermutet man, dass atypische Bakterien den Verlauf von Asthma negativ beeinflussen.
Typische Pneumonien
Bakterielle Erreger sind die häufigsten Verursacher einer Lungenentzündung und meistens wird diese durch das Bakterium Streptococcus pneumoniae verursacht. Andere Bakterien wie z. B. Haemophilus influenzae und Staphylococcus aureus können auch Ursache einer Lungenentzündung sein.
Die Schwere der Lungenentzündung hängt auch von der Art des Erregers ab, weshalb eine Bestimmung des Erregers mittels PCR Panel vorteilhaft ist.
Bordetella pertussis
Der Keuchhusten wird vom Bakterium Bordetella pertussis verursacht. Vor allem Säuglinge und Kleinkinder sind von schweren Krankheitsverläufen betroffen, die in Einzelfällen auch zum Tode führen können. Infektionen mit B. pertussis sind in Österreich meldepflichtig.
In Österreich wurden im Jahr 2024 (Stand: 29.10.2024) bereits 13.441 Bordetella pertussis Infektionen registriert (Link zu Ages). Das ist ein deutlicher Anstieg gegenüber dem Jahr 2023 (2791 Fälle). Im Jahr 2015 waren nur 579 bekannt und während den COVID-19 Pandemie Jahren sogar noch weniger. Mittlerweile kommt es auch bei Erwachsenen gehäuft zu schwereren Verläufen.
Zur Identifizierung von Bordetella pertussis verwendet das Pneumonia-Panel ein Bordetella Insertions-Element (IS) als Target Sequenz, welches in hoher Kopienzahl im Genom vorliegt und daher eine hohe Sensitivität und Spezifität garantiert.
Coxiella burnetti
Coxiella burnetii ist ein kleines gram negatives intrazelluläres Bakterium, das mit Antibiotika behandelt werden kann (z.B. Doxycyclin). Eine akute Infektion beginnt mit Kopfschmerzen, Fieber und Unwohlsein und wird auch Q-Fieber genannt. Die Krankheit klingt nach 2 Wochen gewöhnlich ab, bei älteren und geschwächten Personen kann das Bakterium allerdings Herz, Lunge und Leber angreifen.
Die Übertragung erfolgt nicht über Personenkontakt sondern über Einatmen von infektiösen Aerosolen. Schafe, Rinder und Ziegen sind symptomlose Träger von Coxiella.
Personen die engen Kontakt zu diesen Nutztieren, welche selbst symptomlos sind, haben sind naturgemäß besonders gefährdet. Zusätzlich kann die Krankheit auch durch den Verzehr von infizierter Rohmilch ausgelöst werden.
C. burnetii fühlt sich in Stuhl, Urin, Milch von diesen Nutztieren in landwirtschaftlichen Betrieben wohl. Erwähnenswert ist auch, dass Infektionen über Zecken möglich sind.
Mycobacterium tuberculosis
M. tuberculosis ist das Bakterium, das jährlich weltweit am häufigsten für Todesfälle verantwortlich ist. Obwohl etwa ein Drittel der Weltbevölkerung mit Mycobakterien infiziert ist, ist das Auftreten von Krankheitssymptomen selten. Wie so oft, treten Krankheitsymptome bei immunsupprimierten bzw. geschwächten Personen auf. Die Tuberkulose ist gut mit Antibiotika behandelbar, weshalb aber in Regionen mit schlechtem Gesundheitssystem bzw. Armut die Sterblichkeit bedeutend höher ist. Ein weiteres Problem ist heute, dass viele Behandlungen scheitern, weil sich multiresistente Stämme durch den Missbrauch von Antibiotika gebildet haben.
Infektionen erfolgen meistens über Tröpfchenaufnahme in der Lunge, wobei infizierte Personen das Bakterium über tiefes Ausatmen bzw. Husten weitergeben können.
Aspergillus fumigatus
Aspergillus fumigatus ist ein saprophytischer Pilz, der praktisch weltweit überall vorkommt und in hoher Konzentration in Böden gefunden werden kann.
Aspergillus fumigatus gehört jedoch auch zu den humanpathogenen Pilzen, weil Pilzsporen über die Lunge aufgesaugt werden und der Pilz vor allem in immunsupprimierten Personen zu sogenannten Aspergillomen anwachsen kann.
Gegenwärtig haben die Menschen ein neues Problem geschaffen, weil Azol Wirkstoffe in der Medizin gegen Aspergillosis eingesetzte werden. Da jedoch Azole auch in der Landwirtschaft eingesetzt werden entwickeln Aspergillus Spezies Resistenzen gegen Azole und die medizinische Behandlung wird dadurch unwirksam.
Cryptococcus neoformans & Cryptococcus gattii
Cryptococcus neoformans ist ein hefeartiger Pilz mit weltweitem Vorkommen. Man vermutet, dass Infektionen gewöhnlich über die Lunge durch Einatmen von trockenem Vogelkot erfolgen. Infektionen verlaufen anfangs symptomlos.
Ausgehend von der Lunge können die Pilze jedoch in andere Organe wandern. Erkrankungen treten nach Aktivierung einer latenten Infektion bei Menschen mit Immunschwäche auf, vor allem bei HIV Infektionen oder nach Knochenmarktransplantation. Bei diesen Patienten breitet sich Cryptococcus häufig im Gehirn, der Hirnhaut und der Haut aus. Das kann dann zu Meningitis oder Meningoenzephalitis führen.
Cryptococcus gattii wird in der Umgebung von Bäumen isoliert, vor allem Eukalyptus Bäumen aber auch Koniferen. Während eine durch C. neoformans verursachte Kryptokokkose bei immunsupprimierten Personen mit massiver Immunschwäche auftritt, kann C. gattii auch bei Menschen mit einem intaktem Immunsystem Krankheitssymptome auslösen.
Infektionen werden mit einer Kombination aus Antimykotika behandelt. Die Kombination verhindert Resistenzen und garantiert schnellen Behandlungserfolg.
Pneumocystis jirovecii
Pneumocystis jirovecii ist ein in der Umwelt vorkommender Pilz der zu der Abteilung der Schlauchpilze ( Ascomycota ) gehört. Durch Tröpfcheninfektion kann er die Lunge besiedeln. Bei Menschen mit normalem Immunsystem erscheint eine Infektion symptomlos.
Immungeschwächte Patienten jedoch zeigen Symptome einer Lungenentzündung. Sie leiden unter Fieber, Dyspnoe und einem trockenen Husten
(mehr …)Der vollautomatisierte Extraktionsautomat MagCore Plus II besticht durch seine Aufreinigung in höchster Qualität und der Vielfalt an Kits.
DNA/RNA kann aus einer Vielzahl an Ausgangsmaterialien extrahiert werden: Blut, Plasma, Körperflüssigkeit, Zellen, Gewebe, FFPE, Bakterien, Viren, forensische Proben.
Neben viraler, bakterieller und genomischer DNA erhält man auch spezielle Endprodukte wie zellfreie DNA (ct DNA) oder miRNA (micro RNA).
Und seit 2022 können wir alle Extraktionsautomaten und Kits unter der IVDR Zertifizierung anbieten !
Nachfolgend sehen Sie eine Übersicht der verfügbaren Kits mit Angaben der Kitgrößen und dem validierten Probenmaterial.
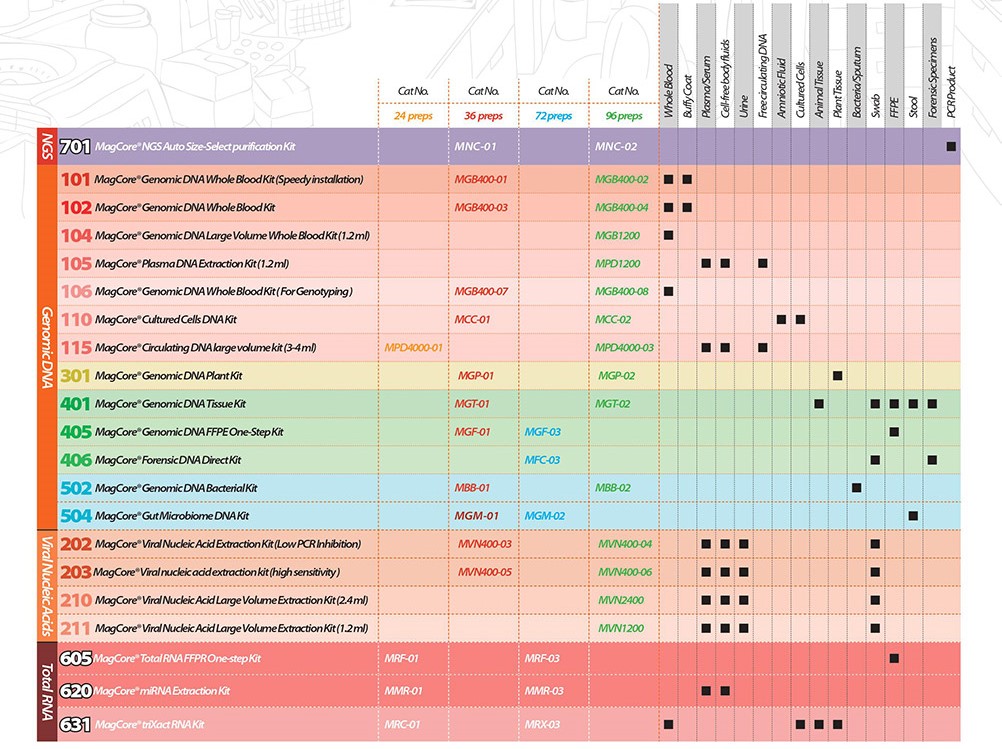
….. und es werden laufend neue Kits entwickelt und das Portfolio erweitert.
Der einzigartige erste vollautomatische Grössenaufreinigungs -Kit für NGS Proben wurde bereits in einem früheren Blog vorgestellt.
Zusätzliche Informationen finden Sie in folgendem Flyer und Video:
In der molekularen Diagnostik werden momentan hauptsächlich Short Read Sequenzierer von den Firmen Illumina bzw. Thermo Fisher eingesetzt weil ihre Technologie sehr verlässlich ist und weniger Fehler produziert als die nächste Generation der Long Read Sequenzierer (PacBio, Oxford Nanopore). Der Nachteil der Short Read Sequenzierungen ist, dass die Vorbereitung relativ aufwendig ist.
Bei der Vorbereitung der Probe, auch "library preparation" genannt, sind mehrere Schritte für eine erfolgreiche Sequenzierung nötig und um qualitativ gute Ergebnisse zu erhalten sollte die library preparation sorgsam und unter höchsten Standards gemacht werden. Folgende Schritte werden benötigt:
Warum brauchen wir eine Größenauswahl ?
–> ein Problem, das Sequenzierer der dritten Generation zu lösen versuchen.
Welche Methoden zur Grössenauswahl gibt es ?
Enzymatisch
z.B Nextera Kits von Illumina. Benutzen Transposons zur Fragmentierung. Ergebnisse aber oft unzufriedenstelllend (Breite Grössenverteilung)
Gel Elektrophorese
Sehr genaue Methode aber extrem aufwendig und zeitintensiv
Magnetic bead – Technologie
DNA-Fragmente der gewünschten Größe binden in Gegenwart des optimierten Puffers an die Beads. Ein Magnet wird verwendet, um die Beads/DNA zu befestigen Kostengünstig, weniger zeitaufwendig als Gel Elektrophorese, gute Qualität
Eigenschaften:
1. Automatische DNA Aufreinigung und Grössenselektion in ~ 30min
2. "magnetic bead" Technologie
3. Hohe Ausbeute

4. Wenig Arbeitszeit
5. Konstant gute Qualität – keine manuellen Fehler
Anwendungen:
1. DNA library peparation für NGS
2. Aufreingungen für PCR, Hybridisierungen oder Real-Time PCR
Digitale PCR ist eine spezielle Weiterentwicklung der klassischen PCR. Das Besondere an dieser PCR Anwendung ist, dass einzelne DNA Moleküle in winzigste Kompartimente getrennt werden, sodass jeweils nur 1 Strang oder keiner in einem Kompartiment amplifiziert wird. Die Art der Aufteilung hängt von der Technologie ab – es wird grob zwischen digital droplet PCR und chamber digital PCR unterschieden. In einem Fall (ddPCR) werden kleinste Öl-Tropfen getrennt und in dem anderem Fall Moleküle in kleinste Mikrobereiche aufgeteilt (cdPCR).
Digitale PCR besticht durch hohe Sensitivität und Spezifität und durch die hohe Verdünnung ist die Beeinträchtigiung durch PCR Inhibitoren fast ausgeschlossen.
Wir sind immer an innovativen Produkten interessiert und können jetzt auch erstmals ein Produkt basierend auf dPCR anbieten.
Die Quantifizierung von BCR-ABL1 bzw. ABL1 aus Vollblut von Patienten mit positiver chronischer myeloischer Leukämie (CML) ist sehr gut geeignet für digitale PCR. Der Dr. PCR™ BCR-ABL1 Major IS Detection Kit ist ein in-vitro-Diagnose Kit und soll das Verhältnis von BCR-ABL1 zu ABL1 messen. Der Kit erlaubt eine akkurate Beobachtung von Leukämie Patienten während einer Therapie bezüglich Verhältnis BCR ABL1 - ABL1 Transkripte.

Der Kit besteht aus 1 Tube dRT-PCR Master Mix und 1 Tube Primer & Probe Mix. Die beiden Reagentien werden in einem Reaktionsgefäß mit dem RNA Extrakt zusammengemischt und in eine mitgelieferte Kartusche injiziert. In der Kartusche wird die Probe in 22000 Kompartimente (50nm) verteilt. Diese Kartusche wird in das real-time Gerät plaziert und nach der qPCR wird das Ergebnis ausgegeben.
Fakten
Ringversuche dienen dazu, die Leistungsfähigkeit eines Labors und ihres Testsystems zu überprüfen. Wenn man medizinische Proben analysiert, muss man sichergehen, dass die Testergebnisse stimmen. Bei Ringversuchen werden identische Proben an teilnehmende Labore verschickt. Es gibt verschiedenste Einflussfaktoren, die die Testergebnisse beeinflussen, wie zum Beispiel die Laborausstattung, die Arbeitsweise der Mitarbeiter und natürlich das Testsystem. Die Testergebnisse werden vom Ringversuch-Anbieter gesammelt und statistisch verarbeitet, um die Messgenauigkeit der Labore zu bewerten
Wir von BioProducts überprüfen unsere Testsysteme mittels Ringversuchen um den Kunden zuverlässige Analysen zu garantieren.
Im Jahr 2022 konnten wir bisher nachweisen, dass unsere HPV Testsysteme den Qualitätsstandards entsprechen. Beim HPV Zervixkarzinom Ringversuch haben wir mit unserem Extraktionssystem von RBC (MagCore® Genomic DNA FFPE One-Step Kit) und dem Panelsystem von Ausdiagnostics (High-Risk HPV Genotyping 8-well (Ref 23201) erfolgreich teilgenommen. Der molekularbiologische Nachweis zur Identifizierung und Unterscheidung der HPV Typen wird immer wichtiger zur Früherkennung von Krebserkrankungen. Es gibt mittlerweile mehr als 120 bekannte HPV Typen und PCR Tests können zuverlässig unterscheiden ob es sich um einen „High Risk“ oder „Low Risk“ Typ handelt. Infektionen mit Hochrisiko-HPV-Typen können zu Tumoren (z. B. Gebärmutterhalskrebs bei HPV16/HPV18) führen, während Niedrigrisiko-HPV-Typen ( z.B. HPV6 und HPV11) für Genitalwarzen verantwortlich sein können.
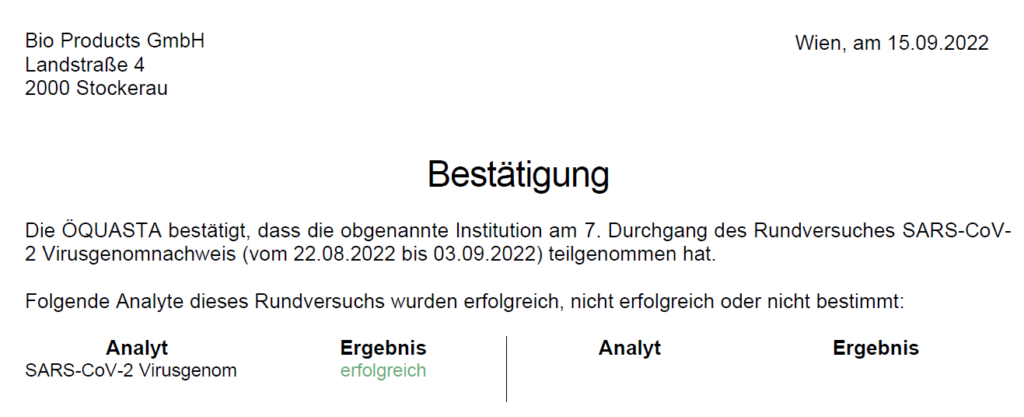
Weiters überprüfen wir regelmäßig die Qualität unserer SARS Cov-2 Testsysteme bei den ÖQUASTA (Österreichische Gesellschaft für Qualitätssicherung und Standardisierung medizinisch-diagnostischer Untersuchungen) Rundversuchen. Sowohl unser POC Analysegerät (LR 1000) als auch das respiratorische Panel (SARS-CoV-2, Influenza & RSV 8-well) von Ausdiagnostics haben 100% der Proben beim letzten Rundversuch (7; August 2022) richtig analysiert. Das gelang nur 73% aller Teilnehmer.
Am 29.09. und 30.09. September 2022 findet die 21. ÖGH (Österreichiche Gesellschaft für Humangenetik) Jahrestagung im Zentrum von Graz statt.
Die ÖGH Jahrestagung ist die wichtigste wissenschaftliche Veranstaltung im Bereich der Humangenetik in Österreich. Die österreichischen Humangenetik-Institute präsentieren die neuesten Trends in der Forschung und Entwicklung mit Schwerpunkt Genom-Diagnostik. Führende Experten werden bei dem Treffen diagnostische Möglichkeiten und Probleme, die die neuesten Technologien in der Genomforschung bringen, diskutieren
Lernen sie die neuesten Errungenschaften in der Humangenetik-Forschung und Diagnostik kennen und besuchen sie in den Pausen unseren Stand. Wir stehen zur Verfügung um mit Ihnen über aktuelle Trends in der Diagnostik zu sprechen und wir bemühen uns Lösungen für offene Fragestellugen anzubieten.
Themen:
Genomsequenzierung, Single-Cell Analysen, Mutationsanalysen, Epigenetik, Tumor und cfDNA, ctDNA und vieles mehr
Wann und wo ?